洛谷P1140 相似基因
题目背景
大家都知道,基因可以看作一个碱基对序列。它包含了44种核苷酸,简记作A,C,G,TA,C,G,T。生物学家正致力于寻找人类基因的功能,以利用于诊断疾病和发明药物。
在一个人类基因工作组的任务中,生物学家研究的是:两个基因的相似程度。因为这个研究对疾病的治疗有着非同寻常的作用。
题目描述
两个基因的相似度的计算方法如下:
对于两个已知基因,例如AGTGATGAGTGATG和GTTAGGTTAG,将它们的碱基互相对应。当然,中间可以加入一些空碱基-,例如:
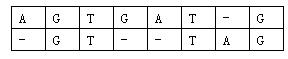
这样,两个基因之间的相似度就可以用碱基之间相似度的总和来描述,碱基之间的相似度如下表所示:
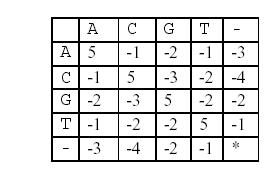
那么相似度就是:(-3)+5+5+(-2)+(-3)+5+(-3)+5=9(−3)+5+5+(−2)+(−3)+5+(−3)+5=9。因为两个基因的对应方法不唯一,例如又有:
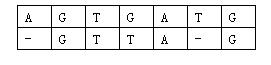
相似度为:(-3)+5+5+(-2)+5+(-1)+5=14(−3)+5+5+(−2)+5+(−1)+5=14。规定两个基因的相似度为所有对应方法中,相似度最大的那个。
输入格式
共两行。每行首先是一个整数,表示基因的长度;隔一个空格后是一个基因序列,序列中只含A,C,G,TA,C,G,T四个字母。1 \le1≤序列的长度\le 100≤100。
输出格式
仅一行,即输入基因的相似度。
输入输出样例
输入 #1
输出 #1

分析
题解来自https://www.luogu.com.cn/blog/zhy137036/ti-xie-p1140-xiang-si-ji-yin-wei-wan-post
动态规划的基本思路,可以分为以下几个步骤:
- 定义状态
- 写出状态转移式
- 根据状态转移式找出递推的顺序
- 处理递推边界
- 找出结果
但是dp的题目还是非常灵活的,还是要自己理解。
对于这道题,最难的就是怎么想出状态转移式。用i,j分别表示以第i个基因结尾的第一个序列和以第j个基因结尾的第二个序列的最大值,通过这个定义设计出dp数组,用两个for循环逐步递推。
要注意的就是边界的设计,dp[0] [0] = 0,表示序列1的第0个基因结尾的序列和序列2的第0个基因结尾的序列最大值,因此只能取0。还有dp[i] [0] 和 dp[0] [j] ,以这种想法进行初始设定。
代码
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
| #include <iostream>
#include <algorithm>
#include <map>
using namespace std;
int value[5][5] = {5, -1, -2, -1, -3, -1, 5, -3, -2, -4, -2, -3, 5, -2, -2,
-1, -2, -2, 5, -1, -3, -4, -2, -1, -0x7fffffff};
map<char, int> gene;
int len1, len2;
char order1[110], order2[110];
int dp[110][110] = {0};
void init();
int main(int argc, const char * argv[])
{
init();
dp[0][0] = 0;
for(int i = 1; i <= len1; i++)
dp[i][0] = dp[i - 1][0] + value[gene[order1[i]]][gene['-']];
for(int j = 1; j <= len2; j++)
dp[0][j] = dp[0][j - 1] + value[gene['-']][gene[order2[j]]];
for(int i = 1; i <= len1; i++)
for(int j = 1; j <= len2; j++)
{
dp[i][j] = max(max(dp[i - 1][j - 1] + value[gene[order1[i]]][gene[order2[j]]],
dp[i - 1][j] + value[gene[order1[i]]][gene['-']]),
dp[i][j - 1] + value[gene['-']][gene[order2[j]]]);
}
cout << dp[len1][len2] << endl;
return 0;
}
void init()
{
gene['A'] = 0; gene['C'] = 1; gene['G'] = 2; gene['T'] = 3; gene['-'] = 4;
cin >> len1;
for(int i = 1; i <= len1; i++)
cin >> order1[i];
cin >> len2;
for(int i = 1; i <= len2; i++)
cin >> order2[i];
}
|





